Team:Goettingen/iGEM deu
From 2012.igem.org
| (One intermediate revision not shown) | |||
| Line 1: | Line 1: | ||
| - | {{GoettingenHeader_deu}} | + | {{GoettingenHeader_deu|deu=Team:Goettingen/iGEM_deu|eng=Team:Goettingen/iGEM}} |
| - | + | == Was ist iGEM? == | |
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
iGEM (International Genetically Engineered Machine competition) ist ein internationaler Wettbewerb, der jährlich vom MIT in Boston, USA veranstaltet wird. Teilnehmen können Studenten aus aller Welt, die sich mit der Molekularbiologie verwandten Themengebieten befassen. Einerseits dient dieser Wettbewerb dazu verschiedene Bildungsaspekte zu kombinieren und soziale Gemeinschaftsarbeit unter Studenten zu fördern und andererseits stellt er eine Sammlung von standardisierten und untereinander auswechselbaren Gen-Bausteinen bereit, welche in lebende Organismen, besonders in Modellorganismen, wie <i>E. coli</i>, eingebracht werden können. | iGEM (International Genetically Engineered Machine competition) ist ein internationaler Wettbewerb, der jährlich vom MIT in Boston, USA veranstaltet wird. Teilnehmen können Studenten aus aller Welt, die sich mit der Molekularbiologie verwandten Themengebieten befassen. Einerseits dient dieser Wettbewerb dazu verschiedene Bildungsaspekte zu kombinieren und soziale Gemeinschaftsarbeit unter Studenten zu fördern und andererseits stellt er eine Sammlung von standardisierten und untereinander auswechselbaren Gen-Bausteinen bereit, welche in lebende Organismen, besonders in Modellorganismen, wie <i>E. coli</i>, eingebracht werden können. | ||
Die teilnehmenden Studenten-Teams werden mit den benötigten Gen-Bausteinen, auch „BioBricks“ genannt, ausgestattet und bearbeiten über mehrere Monate ihr eigenes Forschungsprojekt. | Die teilnehmenden Studenten-Teams werden mit den benötigten Gen-Bausteinen, auch „BioBricks“ genannt, ausgestattet und bearbeiten über mehrere Monate ihr eigenes Forschungsprojekt. | ||
Jedes Jahr wird das als „Registry of Standard Biological Parts“ bezeichnete Register von den mitwirkenden iGEM-Teams um weitere „BioBricks“ erweitert. Diese sind dann in den folgenden Jahren ebenfalls für die iGEM-Teilnehmer zugänglich und können von diesen für ihre eigenen Projekte genutzt werden. | Jedes Jahr wird das als „Registry of Standard Biological Parts“ bezeichnete Register von den mitwirkenden iGEM-Teams um weitere „BioBricks“ erweitert. Diese sind dann in den folgenden Jahren ebenfalls für die iGEM-Teilnehmer zugänglich und können von diesen für ihre eigenen Projekte genutzt werden. | ||
| - | < | + | |
| - | <font | + | <html><center><font size="-2">Das iGEM-Logo. Für die iGEM-Homepage auf das Logo klicken<br> |
| - | Das iGEM-Logo. Für die iGEM-Homepage auf das Logo klicken<br> | + | <a href="https://2012.igem.org"><img src="https://static.igem.org/mediawiki/2012/b/bd/SynBioWiki.png"></a></font></center></html> |
| - | <a href="https://2012.igem.org"><img | + | |
| - | < | + | |
| - | + | ||
Der erste iGEM-Wettbewerb fand im Jahr 2003 statt und war zu diesem Zeitpunkt ausschließlich für Studenten des MIT vorgesehen. In den folgenden Jahren wurde die Teilnahme auch für Teams anderer Universitäten gestattet und im Jahr 2011 traten bereits 165 Teams von Universitäten aus allen Teilen der Welt an. | Der erste iGEM-Wettbewerb fand im Jahr 2003 statt und war zu diesem Zeitpunkt ausschließlich für Studenten des MIT vorgesehen. In den folgenden Jahren wurde die Teilnahme auch für Teams anderer Universitäten gestattet und im Jahr 2011 traten bereits 165 Teams von Universitäten aus allen Teilen der Welt an. | ||
Dieses Jahr nimmt auch ein Team der Universität Göttingen erstmals an diesem Wettstreit teil und sieht diesen als eine großartige Chance zahlreiche Fähigkeiten, die für eine Karriere als Molekularbiologe nötig sind, zu erhalten und zu verbessern. | Dieses Jahr nimmt auch ein Team der Universität Göttingen erstmals an diesem Wettstreit teil und sieht diesen als eine großartige Chance zahlreiche Fähigkeiten, die für eine Karriere als Molekularbiologe nötig sind, zu erhalten und zu verbessern. | ||
Zu diesen Fähigkeiten gehören die Planung und Organisation eines Projektes, einschließlich der Aufstellung eines Teams, Finanzierung der laufenden Ausgaben, Koordination der Laborarbeit, dem Design von Experimenten und der Zusammenarbeit als Team. | Zu diesen Fähigkeiten gehören die Planung und Organisation eines Projektes, einschließlich der Aufstellung eines Teams, Finanzierung der laufenden Ausgaben, Koordination der Laborarbeit, dem Design von Experimenten und der Zusammenarbeit als Team. | ||
| - | Neben der tatkräftigen Arbeit im Labor über die gesamten Sommermonate, sind auch Literatur-Recherchen und eine gut strukturierte Dokumentation der Experimente und derer Ergebnisse von großer Bedeutung. | + | Neben der tatkräftigen Arbeit im Labor über die gesamten Sommermonate, sind auch Literatur-Recherchen und eine gut strukturierte Dokumentation der Experimente und derer Ergebnisse von großer Bedeutung. |
| - | + | ||
| - | + | ||
| - | + | ||
| + | == BioBrick System == | ||
| + | [[File:Biobrick.jpg|250px|thumb|<b>Abbildung 1</b>: Das Prinzip der BioBrick Standardisierung. Um zu der Pubmed-Eintragung der Veröffentlichung zu gelangen, klicken Sie bitte auf die Abbildung. <html><a href="http://www.ncbi.nlm.nih.gov/pubmed/22083734">Mueller KM & Arndt KM (2012). Standardization in synthetic biology. Methods Mol Biol. Vol. 813:23-43.</a></html>]] | ||
| + | Das "BioBrick System" basiert auf der Idee komplexe biologische Systeme in einzelne Komponenten zu unterteilen und diese allen iGEM-Teams zugänglich zu machen. Jede Komponente, auch "BioBrick" oder "Part" genannt, wird von standardisierten Sequenzen flankiert. Dadurch wird es ermöglicht, die Komponenten schnell und problemlos in verschiedenen biologischen Modelorganismen einzufügen. Die standardisierten Sequenzen beinhalten spezifische Restriktionsschnittstellen: für die Enzyme <i>EcoR</i>I und <i>Xba</i>I vor und <i>Spe</i>I und <i>Pst</i>I im Anschluss an die genetische Sequenz eines jeden "Parts" (Abb. 1). Dadurch ist es ebenfalls möglich verschiedene "Parts" miteinander zu kombinieren. Die Restriktionsstellen <i>Spe</i>I und <i>Xba</i>I, die sich zwischen zwei "Parts" befinden, können aneinander ligiert werden und hinterlassen eine sogenannte "Scar" (Narbe). Die Restriktionsschnittstellen werden so miteinander verbunden, dass die beiden Enzyme sie anschließend nicht mehr erkennen können. "BioBricks" werden in der sogenannten "Registry of Standardized Biological parts" eingetragen und z.B. nach Funktion geordnet. Typische "Biobricks" sind Proteine, Promotoren, Primer oder Plasmide. | ||
| - | + | == Part Registry == | |
| - | + | Die sogenannte [[Main_Page|Registry of Standardized Parts] (Eintragung der standardizierten Genbausteine) ist eine online Plattform, die für alle teilnehmenden iGEM-Teams frei zugänglich ist. Jedes Team fügt im Rahmen des Wettbewerbs neue "Parts" in die Datenbank ein und sendet die passenden Genbausteine per Post nach Bosten ins iGEM Hauptquartier. Im Gegenzug wird eine ausgewählte Zusammenstellung, das sogenannte "Distribution Kit", von bereits vorhandenen "Parts" an die teilnehmenden Teams versendet. "Parts", die nicht in der Zusammenstellung enthalten sind, können zusätzlich bei iGEM bestellt werden. Da die Anzahl der teilnehmenden Universitäten am iGEM Wettbewerb jährlich ansteigt, wächst auch die Menge an "Parts" in der Datenbank rapide an. Um die Datenbank übersichtlich und verständlich für neue Teams zu gestalten, wird die Plattform kontinuierlich überarbeitet. So sind umfangreiche Hilfsseiten und Beschreibungen zur Pflege und Eintragung neuer "Parts" zu finden. | |
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | Die sogenannte | + | |
Im Folgenden ist das "Distribution Kit" 2012 und die beinhalteten "Parts" verlinkt: | Im Folgenden ist das "Distribution Kit" 2012 und die beinhalteten "Parts" verlinkt: | ||
| - | + | * [http://partsregistry.org/Help:Distribution_Kits#Using_the_online_QC_resources Distribution Kit 2012] | |
| - | + | * [http://partsregistry.org/assembly/libraries.cgi?id=42 2012 BioBrick DNA Part Repositories 2012] | |
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | |||
| - | |||
| - | |||
| - | |||
{{GoettingenFooter}} | {{GoettingenFooter}} | ||
Latest revision as of 15:27, 21 September 2012
 | |
Deutsch  / English / English  |
 |
Was ist iGEM?
iGEM (International Genetically Engineered Machine competition) ist ein internationaler Wettbewerb, der jährlich vom MIT in Boston, USA veranstaltet wird. Teilnehmen können Studenten aus aller Welt, die sich mit der Molekularbiologie verwandten Themengebieten befassen. Einerseits dient dieser Wettbewerb dazu verschiedene Bildungsaspekte zu kombinieren und soziale Gemeinschaftsarbeit unter Studenten zu fördern und andererseits stellt er eine Sammlung von standardisierten und untereinander auswechselbaren Gen-Bausteinen bereit, welche in lebende Organismen, besonders in Modellorganismen, wie E. coli, eingebracht werden können. Die teilnehmenden Studenten-Teams werden mit den benötigten Gen-Bausteinen, auch „BioBricks“ genannt, ausgestattet und bearbeiten über mehrere Monate ihr eigenes Forschungsprojekt. Jedes Jahr wird das als „Registry of Standard Biological Parts“ bezeichnete Register von den mitwirkenden iGEM-Teams um weitere „BioBricks“ erweitert. Diese sind dann in den folgenden Jahren ebenfalls für die iGEM-Teilnehmer zugänglich und können von diesen für ihre eigenen Projekte genutzt werden.

Der erste iGEM-Wettbewerb fand im Jahr 2003 statt und war zu diesem Zeitpunkt ausschließlich für Studenten des MIT vorgesehen. In den folgenden Jahren wurde die Teilnahme auch für Teams anderer Universitäten gestattet und im Jahr 2011 traten bereits 165 Teams von Universitäten aus allen Teilen der Welt an. Dieses Jahr nimmt auch ein Team der Universität Göttingen erstmals an diesem Wettstreit teil und sieht diesen als eine großartige Chance zahlreiche Fähigkeiten, die für eine Karriere als Molekularbiologe nötig sind, zu erhalten und zu verbessern. Zu diesen Fähigkeiten gehören die Planung und Organisation eines Projektes, einschließlich der Aufstellung eines Teams, Finanzierung der laufenden Ausgaben, Koordination der Laborarbeit, dem Design von Experimenten und der Zusammenarbeit als Team. Neben der tatkräftigen Arbeit im Labor über die gesamten Sommermonate, sind auch Literatur-Recherchen und eine gut strukturierte Dokumentation der Experimente und derer Ergebnisse von großer Bedeutung.
BioBrick System
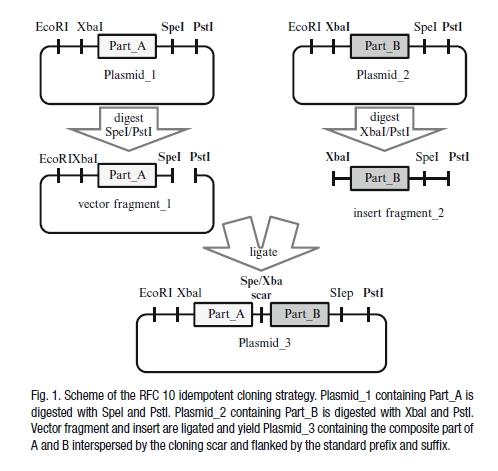
Das "BioBrick System" basiert auf der Idee komplexe biologische Systeme in einzelne Komponenten zu unterteilen und diese allen iGEM-Teams zugänglich zu machen. Jede Komponente, auch "BioBrick" oder "Part" genannt, wird von standardisierten Sequenzen flankiert. Dadurch wird es ermöglicht, die Komponenten schnell und problemlos in verschiedenen biologischen Modelorganismen einzufügen. Die standardisierten Sequenzen beinhalten spezifische Restriktionsschnittstellen: für die Enzyme EcoRI und XbaI vor und SpeI und PstI im Anschluss an die genetische Sequenz eines jeden "Parts" (Abb. 1). Dadurch ist es ebenfalls möglich verschiedene "Parts" miteinander zu kombinieren. Die Restriktionsstellen SpeI und XbaI, die sich zwischen zwei "Parts" befinden, können aneinander ligiert werden und hinterlassen eine sogenannte "Scar" (Narbe). Die Restriktionsschnittstellen werden so miteinander verbunden, dass die beiden Enzyme sie anschließend nicht mehr erkennen können. "BioBricks" werden in der sogenannten "Registry of Standardized Biological parts" eingetragen und z.B. nach Funktion geordnet. Typische "Biobricks" sind Proteine, Promotoren, Primer oder Plasmide.
Part Registry
Die sogenannte [[Main_Page|Registry of Standardized Parts] (Eintragung der standardizierten Genbausteine) ist eine online Plattform, die für alle teilnehmenden iGEM-Teams frei zugänglich ist. Jedes Team fügt im Rahmen des Wettbewerbs neue "Parts" in die Datenbank ein und sendet die passenden Genbausteine per Post nach Bosten ins iGEM Hauptquartier. Im Gegenzug wird eine ausgewählte Zusammenstellung, das sogenannte "Distribution Kit", von bereits vorhandenen "Parts" an die teilnehmenden Teams versendet. "Parts", die nicht in der Zusammenstellung enthalten sind, können zusätzlich bei iGEM bestellt werden. Da die Anzahl der teilnehmenden Universitäten am iGEM Wettbewerb jährlich ansteigt, wächst auch die Menge an "Parts" in der Datenbank rapide an. Um die Datenbank übersichtlich und verständlich für neue Teams zu gestalten, wird die Plattform kontinuierlich überarbeitet. So sind umfangreiche Hilfsseiten und Beschreibungen zur Pflege und Eintragung neuer "Parts" zu finden.
Im Folgenden ist das "Distribution Kit" 2012 und die beinhalteten "Parts" verlinkt:
- [http://partsregistry.org/Help:Distribution_Kits#Using_the_online_QC_resources Distribution Kit 2012]
- [http://partsregistry.org/assembly/libraries.cgi?id=42 2012 BioBrick DNA Part Repositories 2012]
| ↑ Back to top! |

|
 "
"