Team:Osaka/Tests
From 2012.igem.org
Tests
Damage tolerance assay
Radioresistance parts contain codon rarely used in E.coli. Codon optimization and a better expression system are needed to make our parts functional, so we transformed plasmid DNA into E.coli Rosetta.
To measure the DNA damage tolerance conferred by each part, we used DNA damaging agents (such as Mitomycin C and Hydrogen peroxide) as a source of DNA damage and then assayed the survival rates. Transformed E. coli was exposed to DNA damaging agents and then incubated for 2 hours. Cells were plated on agar plates at different dilutions and air dried. Plates were wrapped with aluminum foil and incubated in the dark. Colony-forming units were scored after 16h incubation at 37°C. For detailed protocols, refer to the Protocols page.
The tolerance parts tested were as follows:
Parts containing one gene each
- CDS: [http://partsregistry.org/wiki/index.php?title=Part:BBa_K602005 PprI], [http://partsregistry.org/wiki/index.php?title=Part:BBa_K602006 PprA], [http://partsregistry.org/wiki/index.php?title=Part:BBa_K602007 PprM] or [http://partsregistry.org/wiki/index.php?title=Part:BBa_K602008 RecA]
Parts containing two genes
- CDS1+2: [http://partsregistry.org/wiki/index.php?title=Part:BBa_K602016 PprI+RecA], [http://partsregistry.org/wiki/index.php?title=Part:BBa_K602017 PprA+RecA], [http://partsregistry.org/wiki/index.php?title=Part:BBa_K602020 PprM+RecA], [http://partsregistry.org/wiki/index.php?title=Part:BBa_K602015 PprI+PprA],[http://partsregistry.org/wiki/index.php?title=Part:BBa_K602018 PprI+PprM], [http://partsregistry.org/wiki/index.php?title=Part:BBa_K602019 PprA+PprM],
Discussion
Single-gene parts
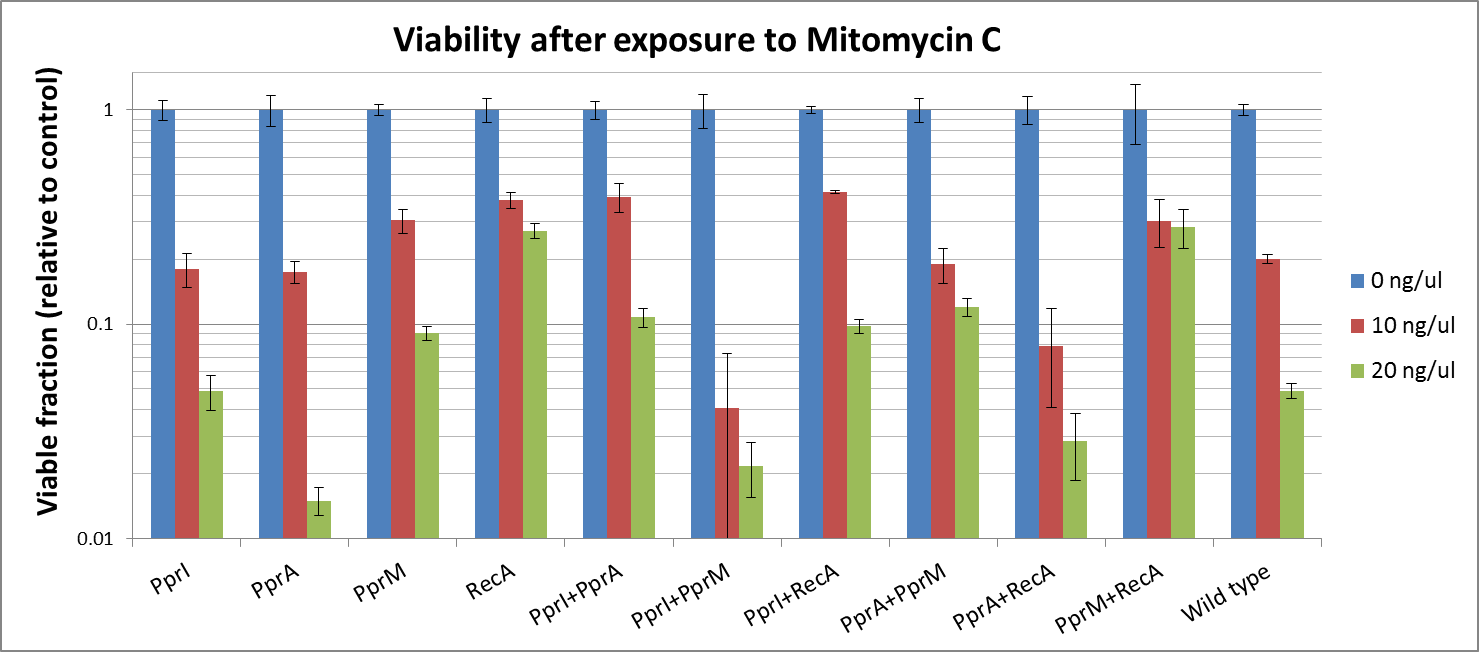
Mitomycine C
- PprI did not appear to increase tolerance, corroborating with its known role as an inducer of other radiotolerance proteins. As E. coli would lack these required D. radiodurans proteins it is expected that PprI would not be able to confer tolerance on its own.
- PprA which repairs blunt-ended breaks, also did not seem to confer tolerance. PprA may not protect against cross-links and single strand breaks induced by Mitomycin C
- Perhaps the most unexpected result was that from PprM. In our previous experiment (using UV irradiation), PprM also appeared to confer a degree of tolerance although it was not significant enough to be of note. This is somewhat in contradiction to literature which describes PprM as a modulator of the PprI-dependent damage response that depends on downstream effector proteins (PprA etc) to carry out its protective role.
耐性を獲得したもの PprM、RecA
singleで耐性の獲得が見られたのは、RecAとPprMの二種類 先行研究によるとRecAはNon-hornologous end-joing repair を行うタンパク質であるとあり、この結果は妥当であるといえよう。特筆すべきは、PprMを導入したE.coliで、統計的に有意義な耐性の獲得が見られたことである。先行研究によると、PprMはDNA修復機能を持つPprAとRecA(後者については意見が分かれる)を誘導する、等の報告があるが、それ自体が修復機能を持つという報告はない。この実験結果について考えられる可能性は二つある。一つはPprMがE.coli固有のRecAの発現を誘導したという可能性。もう一つはPprM自体が何らかの修復能を有しているという可能性である。今後はこの点についてさらに追及(ノックアウト実験とか)をしていく必要性がありそうだ。
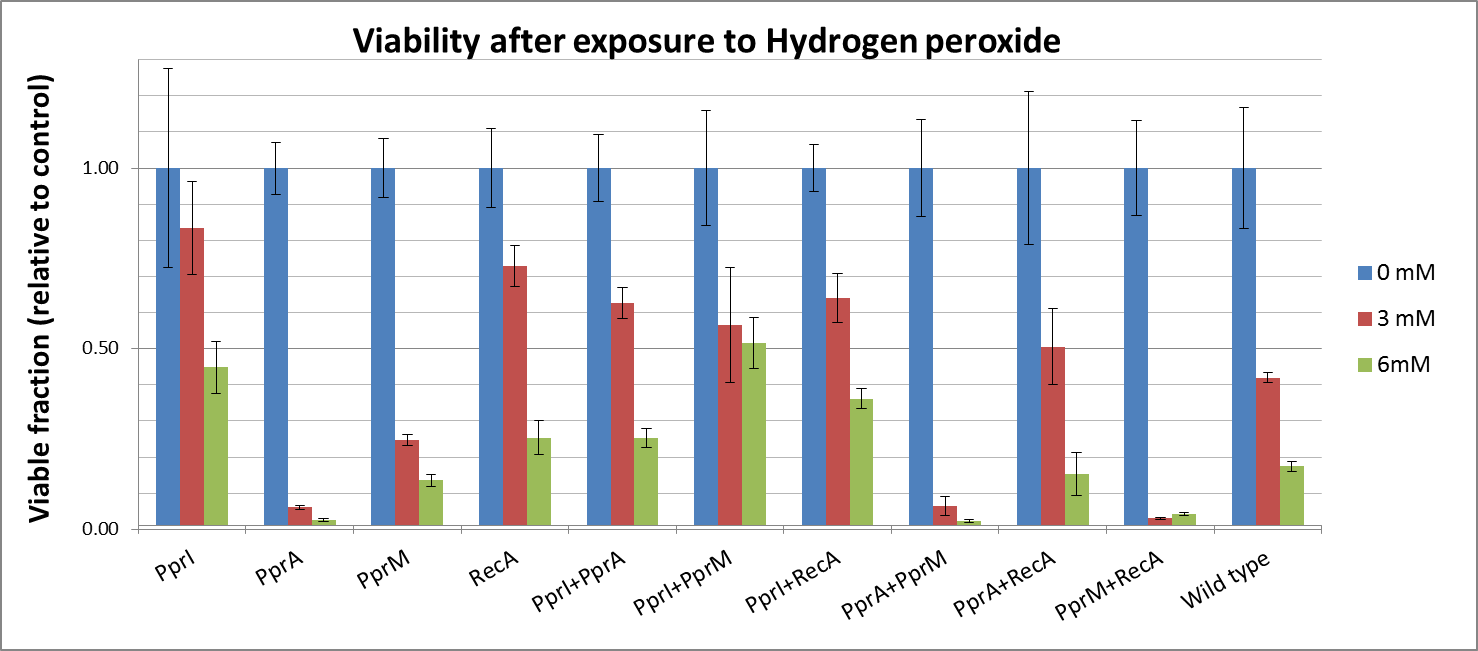
Hydrogen peroxide
singleで耐性の獲得が見られたのは、PprIとRecAの二つ。先行研究によると、PprIは(ほかのRadiotolerance proteinsをInduce するだけでなく)Catalasesを活性化させる作用もある→実験結果は妥当。
Two-gene combinations
Mitomycine C
耐性を獲得したもの combination⇒RecA+PprM、RecA+PprI, PprM+PprA、PprI+PprA combinationで耐性の獲得が見られたのは、PprI+PprA、RecA+PprM、RecA+PprI、PprM+PprAの4つの組み合わせである。このうち、RecA+PprM、RecA+PprI、PprM+PprAの組み合わせについては、singleでも耐性を獲得したRecA、PprMが含まれていることから、さほど、驚くこともない結果といえる。注目すべきは、それぞれ独立では耐性が獲得されなかったPprI+PprAの組み合わせが耐性を獲得したことである。先行研究では、PprIがPprAを誘導し、そしてそのうえで発現したPprAそれ自体がDNA鎖の切断を修復するという(アヤシイ)ことになっていた。この実験結果は、それに反して、PprAが修復能をもつには、PprIの存在が不可欠であるということに、たとえば、DNA修復の際にPprAとPprIが複合体として働くという可能性なども示唆するものであり、極めて重要であるといえる。
Hydrogen peroxide
singleで耐性の獲得が見られたのは、PprIとRecAの二つ。先行研究によると、PprIは(ほかのRadiotolerance proteinsをInduce するだけでなく)Catalasesを活性化させる作用もある→実験結果は妥当。
Conclusion
・MMC→DNA鎖に結合した後、分解され、ラジカル化。DNA鎖切断や架橋形成を引き起こす(遺伝情報を特異的に破壊する。) ・H2O 2→それ自体が反応性が高く、E.coliのあらゆる組織にダメージを与える。(これは、MMCが遺伝情報を特異的に破壊するのと対照的) 仮説
以上の前提を踏まえて、H2O2を用いたTolerance assayでE.coliの生存率を左右したのはH2O2がE.ciliのDNAにダメージをあたえたからというよりも、むしろH2O2がE.coliの諸組織を酸化し、生命機能を維持できなくさせたためだった可能性がある。
根拠
・MMCとH2O2の間の作用機構の相違 ・H2O2を用いたTolerance assay で、(Catalasisを活性化する)PprIをいれたすべてのE.coliについて耐性の上昇がみられた。 ・MMCをもちいたassayにおいて耐性の上昇がみられたPprM、RecA+PprM、PprM+PprA導入体についてをH2O2用いたassayでは耐性の上昇がみられなかったこと。 →BUT、H2O2を用いたassayにおけるRecA singleでの耐性の上昇が説明不可
Promoter assay
We assayed the promoter of the SOS gene RecA ([http://partsregistry.org/wiki/index.php?title=Part:BBa_J22106 J22106]) and sulA ([http://partsregistry.org/wiki/index.php?title=Part:BBa_K518010 K518010]). To measure the DNA damage detection, we used antibacterial agents as a source of DNA damage. To quantitatively and accurately evaluate the promoter activity, dual luciferase assay method was employed. Transformed E. coli was exposed to antibacterial agents and then incubated for 2 hours. For details check the Protocols page.
We were going to assemble the Dual Luciferase Assay System from existing Biobrick parts, but did not make it.
 "
"